Busca por Possíveis Antígenos para Bactérias Super-Resistentes

A Revista Eletrônica PesquisABC possui o seguinte registro ISSN: 2675-1461
Larissa Almeida Brigagão a , Ana Paula Mattos Arêas b*
a Centro de Ciências Naturais e Humanas, Universidade Federal do ABC (UFABC), Brasil
b Centro de Ciências Naturais e Humanas, Universidade Federal do ABC (UFABC), Brasil
*e-mail do autor de correspondência: Este endereço de email está sendo protegido de spambots. Você precisa do JavaScript ativado para vê-lo.
ORCID ID de Ana Paula Mattos Arêas (Arêas APM): https://orcid.org/0000-0002-1946-9285
Resumo: Bactérias super-resistentes já se mostravam um problema crescente para a saúde mundial, após a pandemia do COVID-19, quando foi observado um aumento dos casos e uma preocupação ainda maior devido ao uso indiscriminado de antimicrobianos. O presente projeto busca maneiras de encontrar possíveis antígenos para o desenvolvimento de medidas profiláticas e terapêuticas contra bactérias de importância médica que formam o grupo ESKAPE, utilizando ferramentas de bioinformática para análise de transportadores do tipo ABC, analisando regiões específicas da proteína, por meio da comparação das sequências com Bancos de Dados de proteínas, de domínio público. Também foram realizadas análises de características físico-químicas, análise quanto aos domínios funcionais e estrutura, além de uma análise teórica de imunogenicidade. O projeto foi finalizado com 33 proteínas que apresentam características positivas para a composição de novas terapias.
Palavras-chave: bactérias ESKAPE, transportadores do tipo ABC, vacinas
Abstract: Highly resistant bacteria have already proven to be a growing concern for global health. Following the COVID-19 pandemics, there has been an increase in cases and even greater concern due to the indiscriminate use of antimicrobials. This project aims to identify potential antigens for the development of prophylactic and therapeutic measures against bacteria of medical relevance, which belongs to the ESKAPE group, using bioinformatics tools to analyze ABC transporters, focusing on specific protein regions, by sequence comparison using public domain-protein database. Additionally, physical-chemical characteristics, functional domains and structural analysis were conducted, along with an immunogenicity analysis. The project concluded with the identification of 33 proteins exhibiting positive characteristics for the development of new therapies.
Keywords: ESKAPE bacteria, ABC transporters, vaccines
Nos últimos anos, o desastre na gestão da resposta à pandemia de COVID-19, que resultou no alastramento do vírus SARS-CoV2 em diversas faixas da população trouxe à tona outros efeitos colaterais. O uso inadequado de antimicrobianos sem eficácia comprovada no tratamento de COVID, como a azitromicina, somado ao crescente uso indiscriminado de outros antibióticos no tratamento de doenças bacterianas descortinou a fragilidade das terapias hoje existentes. A consequência direta destas ações é a maior frequência das mutações, que por sua vez, geram uma das maiores vantagens desses microrganismos contra os tratamentos existentes: a super resistência. Os antibióticos acabam por eliminar as bactérias mais suscetíveis a ele, isto é, bactérias mais fracas em relação àquele medicamento, contudo, as bactérias mais fortes acabam ficando no organismo e se multiplicando, formando outras bactérias fortes, como pode ser visto na imagem 1.
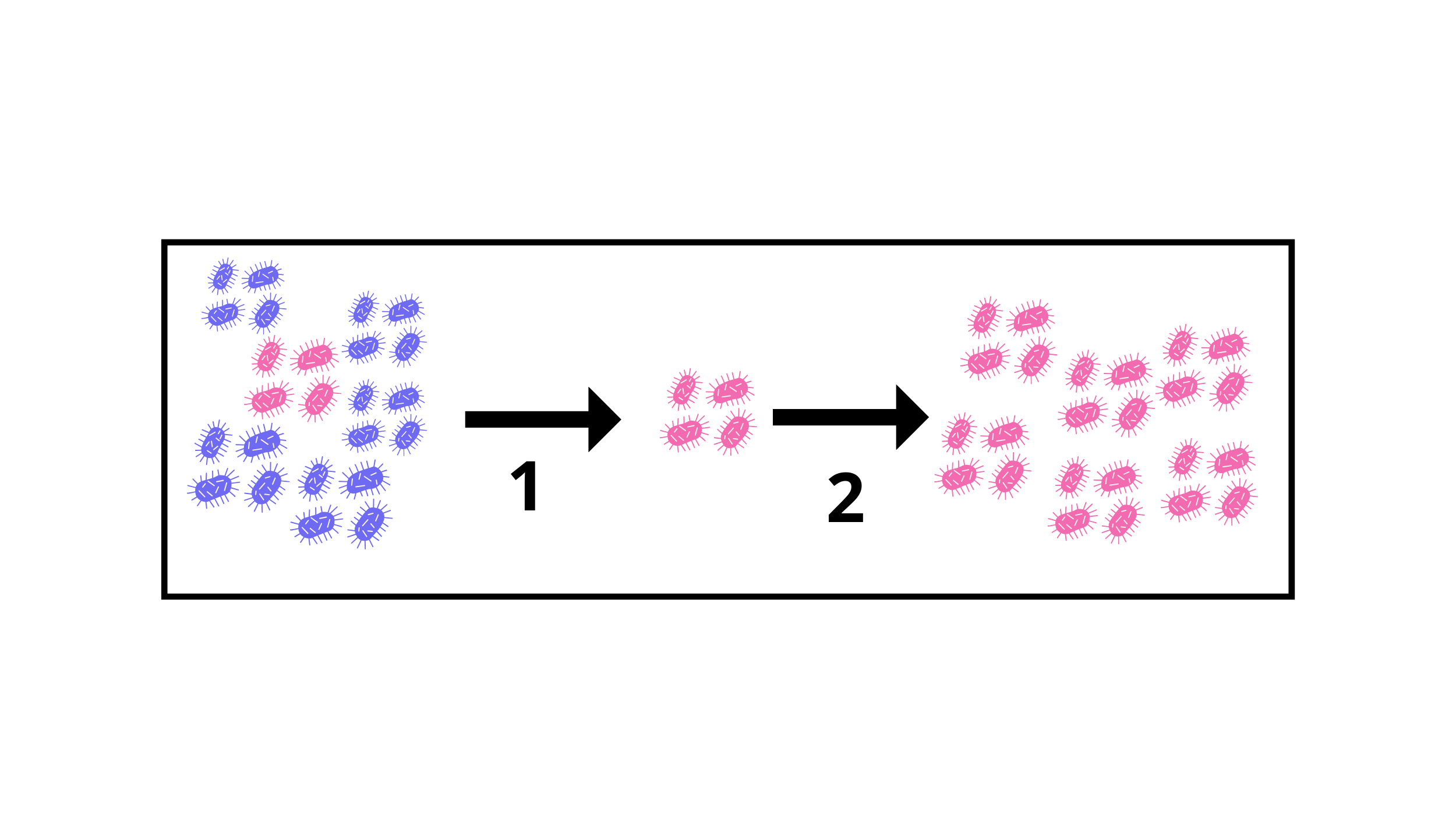
Figura 1. A figura simplifica o processo de resistência bacteriana, em 1 indica a utilização de um antibiótico o qual as bactérias azuis, suscetíveis a esse antibiótico, morrem, restando apenas as bactérias rosas que são resistentes ao agente. Em 2, observamos a reprodução das bactérias rosas por bipartição, processo em que 1 célula forma 2 idênticas. Uma vez que as bactérias mais fracas morreram, as mais fortes podem se estabelecer mais facilmente, não tendo disputa por nutrientes ou espaço, aumentando assim a quantidade de bactérias resistentes.
Dentro do conjunto das bactérias super-resistentes, pode ser ressaltado o grupo denominado por ESKAPE, formado pela Enterococcus faecium, Staphylococcus aureus, Klebsiella pneumoniae, Acinetobacter baumanni, Pseudomonas aeruginosa e espécies do gênero Enterobacter. Essas bactérias circulam nos hospitais e são os responsáveis por muitos dos conhecidos diagnósticos de “infecção hospitalar” de pacientes que já possuem comorbidades (doenças preexistentes que podem agravar a infecção). A fim de encontrar novos tratamentos para esse grupo de patógenos foi proposta a busca por possíveis antígenos a partir de um grupo de proteínas presentes nesses organismos.
A princípio, o que seria um antígeno? Em outras palavras, um antígeno é uma substância (na maioria das vezes, uma proteína), que pode ser microbiana ou não, que é reconhecida pelo sistema imunológico, como sendo algo estranho, algo que não faz parte da sua composição própria. Portanto, sempre que um microrganismo entra em contato com o seu corpo, alguma substância dele será reconhecida por células específicas suas, essas células possuem diversos receptores (funcionam como antenas/sensores) que conseguem reconhecer uma gama enorme de antígenos. Esses antígenos são compostos por regiões específicas chamadas de epítopos, que são as partes às quais um anticorpo, uma célula do sistema imune, reconhece um antígeno. Em outras palavras, não é o antígeno inteiro que é reconhecido, mas estas partes específicas (epítopos), que dizemos então que são mais imunogênicas. Estas são as regiões mais interessantes para compor vacinas. Por isso, alguns tipos de vacinas trabalham simulando os epítopos de microrganismos.
Cientistas buscam as regiões que seriam reconhecidas em uma infecção tradicional, transformam em uma formulação terapêutica com outros tipos de compostos para gerar estabilidade, melhorar o reconhecimento dos antígenos e aplicam nos indivíduos. A partir daí, o organismo reconhece aquela região e cria uma memória imunológica, garantindo com que o sistema imunológico seja mais eficiente durante a infecção propriamente dita.
Nesse projeto, foi escolhido um grupo de proteínas como fonte de possíveis antígenos para estudo: os denominados Transportadores do tipo ABC, que são extremamente importantes para a sobrevivência de microrganismos, servindo como exportadores e importadores de nutrientes e outros componentes de manutenção à vida. Os transportadores ABC são compostos por 3 tipos de proteínas, sendo as escolhidas para o projeto as que se ligam diretamente à substância transportada naquele sistema. Também foram selecionados somente sistemas ABC de importação de nutrientes. As proteínas de interesse estão localizadas na membrana da bactéria, por estarem mais expostas no microrganismo, portanto mais acessíveis para o reconhecimento de um anticorpo, por exemplo. Na imagem de número 2 é possível observar os conceitos abordados neste estudo.
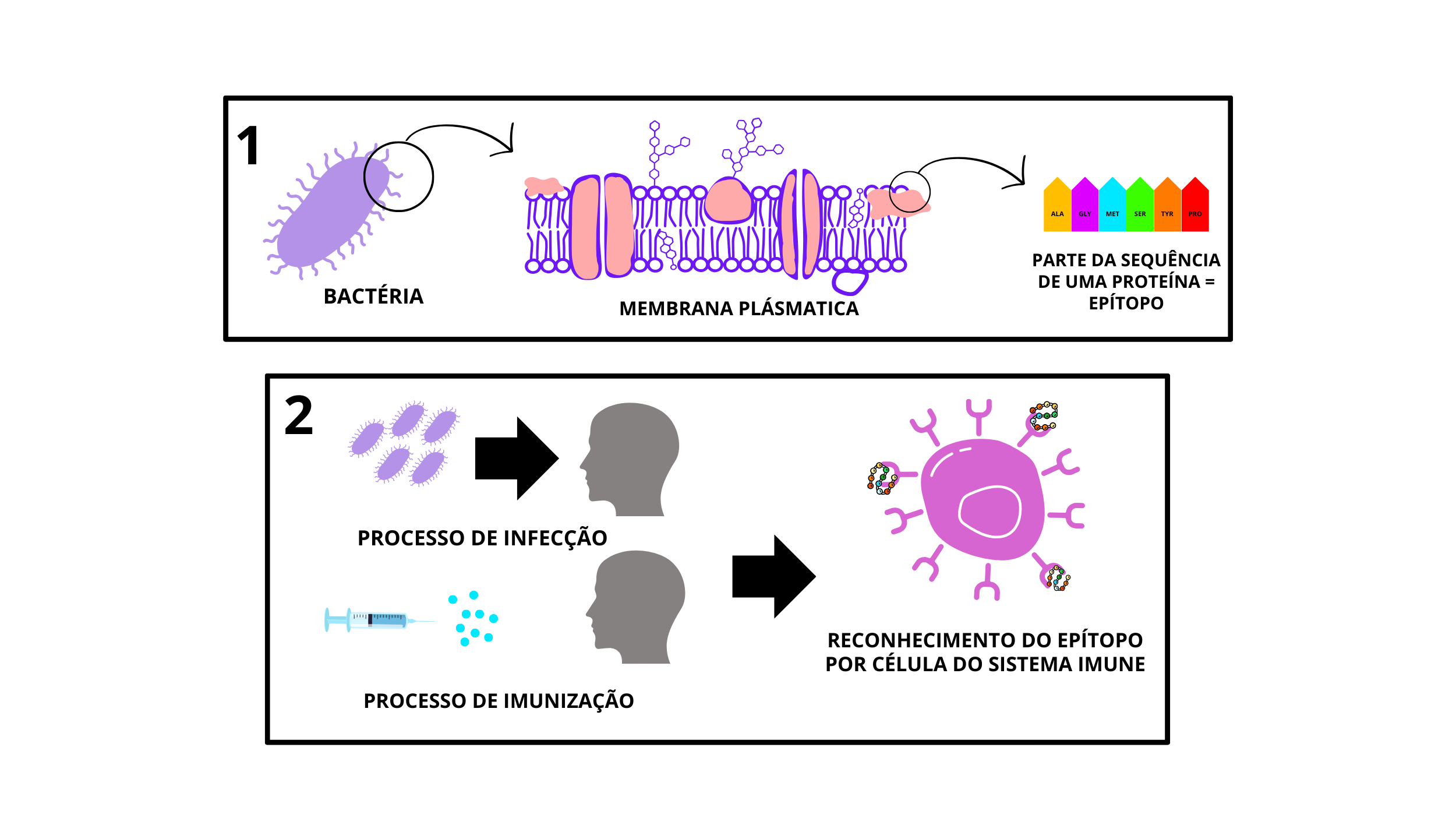
Figura 2. No quadro de número 1 pode-se observar uma bactéria, em seguida um zoom em sua membrana plasmática formada por lipídeos e proteínas, representadas em rosa, e a partir do zoom de uma proteína ancorada na membrana, observa-se uma sequência de aminoácidos que representa um epítopo. Em 2, observa-se o reconhecimento de um antígeno por um anticorpo por meio de dois tipos de processo, um naturalmente, devido a uma infecção, e outro de forma artificial, por meio do processo de imunização, ambos gerando memória e resposta imunológica.
Foram encontrados os epítopos do grupo ESKAPE, utilizando proteínas do tipo Transportadores ABC de membrana. Foram utilizadas ferramentas de bioinformática amplamente utilizadas hoje para, além de poupar recursos, acelerar as etapas da pesquisa.
Inicialmente, as proteínas foram selecionadas utilizando o Uniprot, uma base de dados com anotações de proteínas de diversos organismos, que já foram sequenciadas por outros cientistas. Nessa etapa, foi preferível a busca por proteínas que fossem Transportadores ABC de importação e que estavam localizadas na membrana plasmática, como já dito anteriormente. Um outro ponto muito importante era selecionar proteínas de superfície da célula bacteriana que não atravessassem a membrana. Proteínas deste tipo (transmembrana) são um desafio biotecnológico para produção em laboratório. Desta forma, estar na superfície era o critério de inclusão e, atravessar a membrana, o de exclusão. Também foi dada preferência para a substância transportada pelo sistema analisado, optando-se por ligantes de metais, que são nutrientes essenciais para o crescimento bacteriano.
Foram selecionadas 10 proteínas para cada bactéria do grupo, totalizando 60 proteínas. Em seguida, foram utilizadas ferramentas para verificar se essas proteínas estavam realmente localizadas na membrana das bactérias. Foi percebido que as anotações disponibilizadas não eram totalmente consistentes e misturavam proteínas transmembrana e outras com funções variadas, logo que não eram interessantes para essa pesquisa. Das 60 proteínas, apenas 27 correspondiam aos critérios mencionados e ainda assim, com um certo grau de incerteza. Para resolver esse problema, foi alterado o método de pesquisa no Uniprot, especificando ainda mais o substrato de ligação, ou seja, o elemento que àquela proteína se liga, o que daria mais certeza que era uma proteína de membrana. A partir disso, foram incluídas 6 proteínas para análise cuja anotação se referia, com um grau de certeza bem alto, a um transportador de metal sem domínio transmembrana.
Com a seleção das 33 proteínas, a próxima etapa teve como base o alinhamento (comparação de sequências) das mesmas com outras existentes utilizando o BLASTp, uma ferramenta que faz a comparação entre as proteínas de interesse com aquelas que estão no banco de dados, permitindo verificar o quanto essas proteínas estão conservadas dentro do mesmo grupo ESKAPE e em outros organismos também. Essa etapa é muito importante para verificar se um antígeno será eficaz para a bactéria em questão, se ele está presente em outras sequências da mesma espécie e de espécies diferentes, não sendo, portanto, uma exceção naquele organismo específico o qual foi encontrado, além de não ser reativo para o organismo humano (o hospedeiro ao qual se destina a futura formulação vacinal). Encontrou-se que a maioria das proteínas está bem conservada dentro do grupo ESKAPE. Isso quer dizer que a proteína ou uma sequência similar aparece nas bactérias estudadas, o que indica que poderia ser um antígeno não só para a espécie descrita, mas também para outras bactérias do grupo, um resultado excelente.
Em seguida, foram analisadas algumas características físico-químicas da proteína, por ferramentas disponíveis no portal proteômico Expasy, como o ponto de fusão e o nível de instabilidade. Esses parâmetros são importantes para quando essas proteínas forem produzidas em laboratório. Verificou-se também os domínios de cada uma, que são regiões com funções específicas, como regiões de ligação com metais, regiões responsáveis por proteção, entre outras, utilizando o programa Pfam e o banco de dados do NCBI. Foram encontradas também as estruturas secundárias formadas pelas proteínas, que podem estar estritamente relacionadas com seus domínios e funções. Além disso, realizou-se a predição da estrutura do RNA mensageiro das proteínas, utilizando-se os softwares Jpred4 e RNAdraw. Por fim, buscou-se também as possíveis regiões exatas em que pode acontecer a ligação entre o epítopo e o anticorpo, conhecidas como as regiões de ligação de MHC, sendo de classe I ou de classe II, regiões extremamente importantes para a identificação de um epítopo. Quando o sistema imunológico capta um antígeno, ele o processa e os seus fragmentos (os epítopos) são complexados ao MHC – seja de classe I ou II, cuja diferença não será detalhada aqui. O complexo MHC-epítopo vai para a superfície de um tipo específico de célula chamada de célula apresentadora de antígeno. Uma vez exposto na superfície, o complexo pode ativar células T, iniciando a resposta imunológica adaptativa, que em última instância, gera a proteção àquele patógeno e a memória imunológica. Portanto, duas premissas precisavam ser cumpridas para as proteínas serem bons antígenos: 1. Estarem expostos na superfície, para serem acessíveis ao arsenal imunológico do hospedeiro; 2. Seus epítopos serem capazes de se ligar ao MHC para iniciar a resposta imunológica, etapa essencial para promover resposta adaptativa e memória.
Após a finalização de todas as análises descritas, observou-se que as 33 proteínas que foram selecionadas, apesar de nem todas apresentarem as características desejadas inicialmente de serem ligantes de metais, possuem alto potencial de serem antígenos, devido às suas características físico-químicas, seus domínios, sua localização e por apresentarem regiões possíveis de ligação de MHC. E agora, quais são os próximos passos?
Ainda existem muitas análises que podem ser feitas por bioinformática, como realizar a modelagem dessas proteínas, que consiste em calcular e visualizar as estruturas em 3D dessas proteínas, além de outras análises em laboratório que podem apresentar desafios ao projeto, como a dificuldade da expressão das proteínas, ou seja (produção em meio utilizando DNA recombinante). Contudo, nossos resultados indicam a possibilidade da construção de uma proteína quimérica, que é uma nova proteína com pedacinhos de várias, essa proteína poderia servir como antígeno para mais de uma bactéria do grupo ESKAPE, o que seria extremamente eficiente já que muitos estudos indicam que existe uma frequência de coinfecção com bactérias do grupo em um mesmo indivíduo. Nossos resultados são animadores, cumprem com a nossa proposta e reconhecemos que ainda há muito o que ser explorado.
Referências
Organização Pan-Americana da Saúde. Americas report surge in drug-resistant infections due to misuse of antimicrobials during pandemic. Disponível em: https://www.paho.org/en/news/17-11-2021-americas-report-surge-drug-resistant-infections-due-misuse-antimicrobials-during. Acesso em: 01/04/2022.
Santos Zonta, F. do N. et al. Colonization by ESKAPES and clinical characteristics of critically ill patients. Global Illness 19, 214–254 (2020).
Mulani, M. S. et al. Emerging Strategies to Combat ESKAPE Pathogens in the Era of Antimicrobial Resistance: A Review. Front. Microbiol. 10, 539 (2019). https://doi.org/10.3389/fmicb.2019.00539
Higgins, C. F. ABC transporters: physiology, structure, and mechanism – an overview. Res. Microbiol. 152, 205–210 (2001).
Tong, J. C. & Ren, E. C. Immunoinformatics: current trends and future directions. Drug Discov. Today 14, 684–689 (2009). https://doi.org/10.1016/j.drudis.2009.04.001
Roitt, I. M., Male, D. K. & Brostoff, J. Fundamentos de Imunologia (12ª ed.). Elsevier (2011).

Redes Sociais