Investigando as mutações presentes no DNA do Citomegalovírus Humano

A Revista Eletrônica PesquisABC possui o seguinte registro ISSN: 2675-1461
Giullia Liss Monteiro Alves*, Maria Cristina Carlan da Silva**.
* Discente de graduação pela Universidade Federal do ABC (UFABC)
ORCID ID: 0009-0003-4243-5635
** Docente de graduação na Universidade Federal do ABC (UFABC)
Resumo: O Citomegalovírus Humano (HCMV) é um tipo de Herpes vírus que pode infectar somente seres humanos. Em indivíduos com o sistema imunológico comprometido, pode causar doenças graves e, até, risco de vida. O HCMV possui a capacidade de infectar o hospedeiro e permanecer nele durante toda a sua vida, pois desenvolveu estratégias que utilizam diversas proteínas virais para inibir a proteção imunológica do indivíduo. Essa pesquisa teve como objetivo investigar a presença de mutações na região do DNA do HCMV responsável pela produção dessas proteínas virais capazes de driblar o sistema imunológico. Essas mutações foram verificadas em amostras de DNA de dez pacientes que receberam transplante de rim pelo Hospital do Rim (HRim/São Paulo) e são positivos para o HCMV. Foi possível identificar diferentes mutações. Entretanto, foi identificada uma mesma variação de A para G no nucleotídeo 299 ou 302 (devido à inserção de uma trinca em uma região anterior) da sequência, que levou à mudança do aminoácido 75 de Metionina para Valina. Este estudo inicial aponta a necessidade de mais pesquisas para verificar se as mutações podem causar alterações na função das proteínas virais e resultar em alguma mudança na doença e no quadro clínico do paciente.
Palavras-chave: citomegalovírus humano (HCMV); UL111A; cmvIL10; LAcmvIL10.
Abstract: Human Cytomegalovirus (HCMV) is a type of Herpes virus that can only infect humans. In immunocompromised individuals, it can cause serious and even life-threatening illnesses. HCMV has the ability to infect the host and remain there throughout its life, as it has developed strategies that use several viral proteins to inhibit the individual's immunological protection. This research aimed to investigate the presence of mutations in the region of HCMV DNA responsible for the production of these viral proteins capable of evading the immune system. These mutations were verified in DNA samples from ten patients who received a kidney transplant at Hospital do Rim (HRim/São Paulo) and are positive for HCMV. It was possible to identify different mutations. However, the same variation from A to G was identified at nucleotide 299 or 302 (due to the insertion of a triplet in a previous region) of the sequence, which led to the change of amino acid 75 from Methionine to Valine. This initial study highlights the need for more research to verify whether mutations can cause changes in the function of viral proteins and result in any changes in the disease and the patient's clinical condition.
Keywords: human cytomegalovirus (HCMV); UL111A; cmvIL10; LAcmvIL10.
Tá, mas o que é Citomegalovírus e por que devemos estudá-lo?
O Citomegalovírus Humano (HCMV) é um tipo de Herpes vírus que ataca apenas seres humanos e provoca a produção de células grandes, chamadas células citomegálicas, daí o nome “Citomegalovírus". A infecção por HCMV é muito comum e prevalente no mundo inteiro1. Em indivíduos saudáveis, a infecção causa sintomas leves, comuns em muitos outros tipos de doenças, como mononucleose. Já em indivíduos com o sistema imunológico comprometido, a infecção é grave e apresenta risco de vida1. Após transplante de órgão, o HCMV é a principal causa de complicações infecciosas1, pois o vírus possui a capacidade de infectar o paciente e permanecer em seu organismo durante toda a sua vida. Assim, quando o sistema imunológico está fortalecido, o vírus fica “adormecido”, não podendo infectar outras pessoas; porém, quando o sistema imunológico está debilitado, o vírus pode ser “ativado”, podendo infectar outras pessoas1 e resultando em risco para o indivíduo, pois o vírus pode gerar doenças mais graves.
Mas, afinal, como o vírus faz isso? Esse vírus adquiriu a capacidade de “sequestrar” e “explorar” uma proteína, que chamamos de IL-102. Essa proteína é produzida pelas células humanas e possui propriedades anti-inflamatórias e imunossupressoras, ou seja, ela diminui a resposta imunológica3. Sabe quando você está com uma dor de garganta e há uma produção exagerada de pus? A IL-10 é a proteína responsável por diminuir essa resposta exagerada do sistema imunológico, levando à diminuição da produção desse pus. Essa proteína é produzida a partir de um gene, ou seja, de uma região do nosso DNA. O vírus incorporou ao seu DNA esse gene, chamado de UL111A, responsável pela produção de oito proteínas4 5 6, sendo que duas delas são as mais estudadas. Elas se chamam: (i) cmvIL-104, também chamada vIL-10A; e (ii) LAcmvIL-105, também chamada vIL-10B. Essas duas proteínas possuem funções parecidas com a IL-10 e, assim, o vírus consegue driblar o sistema imunológico e não ser eliminado, permanecendo no indivíduo por toda a sua vida2.
Diante disso, questões vieram à tona: Há mutações na região UL111A do DNA do vírus? Onde estão localizadas essas mutações? Elas podem alterar a função das proteínas produzidas a partir dessa região? Poucos estudos analisaram a presença de mutações na parte UL111A do Citomegalovírus Humano em amostras de indivíduos infectados por esse vírus. Os estudos feitos demonstraram mutações diferentes em diversas linhagens do vírus7 8 9 10. Assim, são necessários mais estudos para analisar as mutações presentes e seu impacto no quadro clínico do paciente. Daí que essa pesquisa busca investigar a presença de mutações na região UL111A do HCMV em amostras de dez pacientes que receberam transplante de rim pelo Hospital do Rim e que, por essa condição, estão com o sistema imunológico comprometido.
Antes de continuar, você sabe o que é DNA, nucleotídeo, aminoácido e proteína?
Para entender melhor esse estudo, é preciso compreender alguns desses conceitos. O DNA é o código genético. Por meio dele, as informações biológicas são armazenadas e é possível produzir as moléculas que o corpo necessita. O DNA é composto por duas fitas, como dois barbantes, que se ligam entre si por ligações de hidrogênio. As fitas são formadas pela ligação de nucleotídeos. Os nucleotídeos são compostos por três partes: uma pentose, que é um açúcar; uma base nitrogenada; e um grupo fosfato. O DNA possui quatro tipos de bases nitrogenadas: a Timina (T), a Adenina (A), a Citosina (C) e a Guanina (G). Na Figura 1, há quatro tipos de nucleotídeos presentes no DNA. Eles se diferem apenas pela base nitrogenada que possuem. É possível perceber que, por meio da ligação de hidrogênio, cada base nitrogenada se liga a uma determinada base (A só se liga com T e C só se liga com G). Assim, se há uma mutação, ou seja, uma variação de base nitrogenada, outra base, complementar a essa, se ligará a essa base mutada, resultando em uma mutação, ou seja, em uma troca do par de bases.
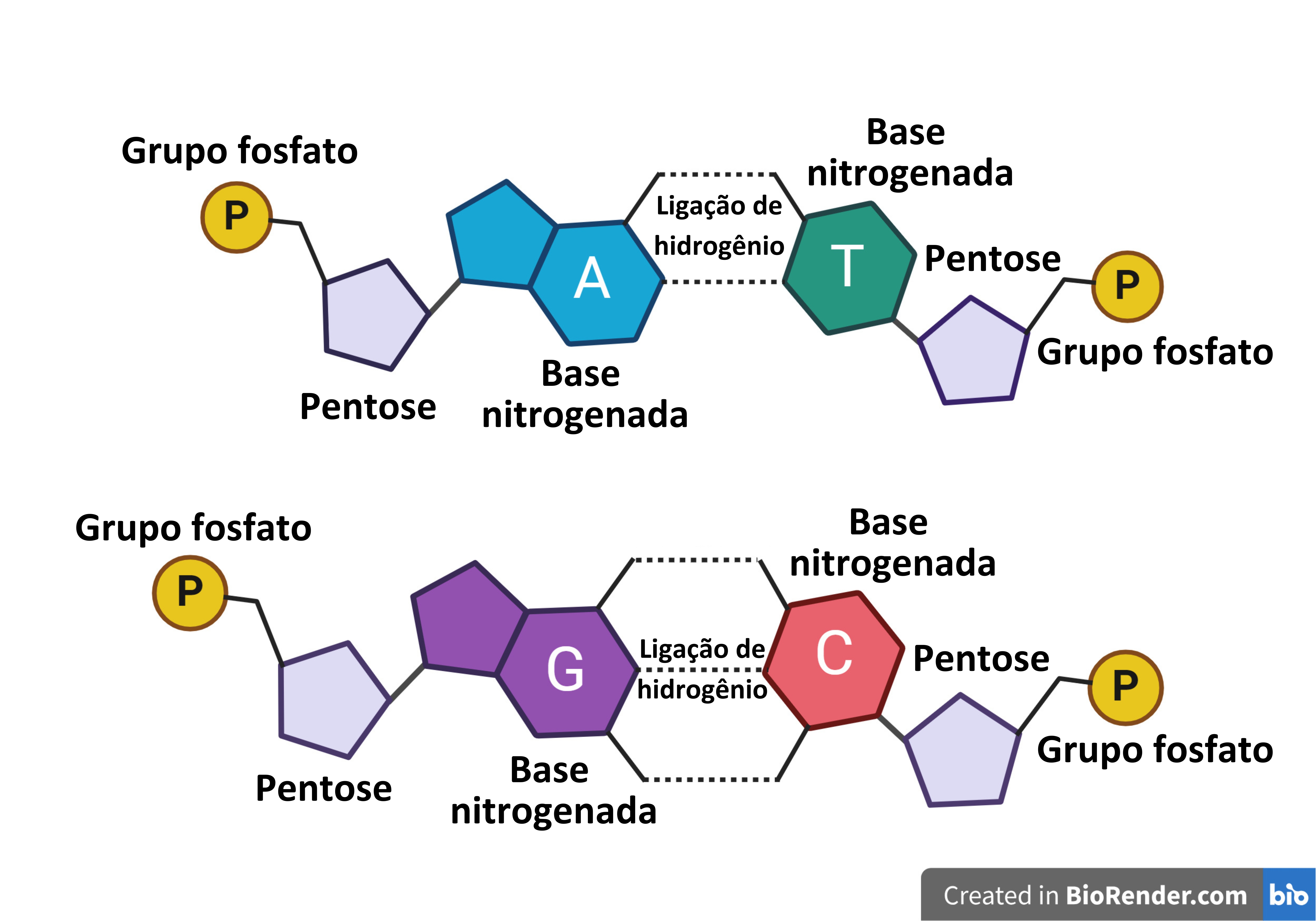
Figura 1. Simplificando o DNA. Elaborado pelas autoras por meio do Bio Render e do Canva.
Para a produção de proteínas, os genes, ou seja, as regiões do DNA, são transcritos, ou seja, copiados, em um RNA, chamado RNA mensageiro (mRNA). O RNA é composto por apenas uma fita e é formado pela ligação de nucleotídeos. Este mRNA é usado para a produção de proteínas nos ribossomos da célula. O ribossomo é uma organela, ou seja, um “órgão” da célula, responsável pela produção de proteínas. Essa produção é feita a partir da combinação de três nucleotídeos, que são lidos pelo ribossomo e correspondem a um aminoácido. Dependendo das bases nitrogenadas e da ordem desses nucleotídeos, um aminoácido diferente é formado. Ao todo, há vinte tipos de aminoácidos, sendo eles: alanina, arginina, aspartato, asparagina, cisteína, fenilalanina, glicina, glutamato, glutamina, histidina, isoleucina, leucina, lisina, metionina, prolina, serina, tirosina, treonina, triptofano e valina.
O conjunto dos três nucleotídeos que formam um aminoácido é chamado de códon. O stop códon, ou códon de parada, é a combinação de três nucleotídeos que não formam nenhum aminoácido, fazendo com que a produção de proteínas seja interrompida. Uma sequência de aminoácidos ligados corresponde a uma proteína. Assim, como a sequência de nucleotídeos interfere na produção de diferentes aminoácidos, a sequência de aminoácidos difere cada uma das proteínas.
E agora, como encontrar os resultados?
Para alcançar o objetivo dessa pesquisa, foi necessária a realização de alguns passos. Em primeiro lugar, o DNA dos dez pacientes passou por um procedimento denominado PCR em Tempo Real, para verificar se esses pacientes realmente eram positivos para o HCMV. Depois que todos testaram positivo, essas amostras passaram por outro processo chamado Nested PCR, no qual o DNA é amplificado, ou seja, se há uma quantidade pequena de DNA, após isso, a quantidade é aumentada. No caso desse estudo, esse aumento foi de 25 vezes. Com essa amostra aumentada, ocorreu a purificação desse DNA, ou seja, tudo o que não era DNA foi descartado. O DNA purificado foi enviado ao Instituto Pasteur de São Paulo e foi sequenciado, ou seja, com esse procedimento, foi possível saber a ordem das bases nitrogenadas do DNA e, assim, montar a sequência. Com essa sequência de “A”s, “T”s, “C”s e “G”s, foi possível comparar a região UL111A de uma linhagem de laboratório do Citomegalovírus, chamada de LISA, que já é conhecida e que foi retirada do GenBank (um banco de dados que armazena sequências de nucleotídeos e proteínas de DNA conhecidos), com a região UL111A de cada um dos dez pacientes e verificar as mutações presentes. Os passos mencionados foram organizados na Figura 2.
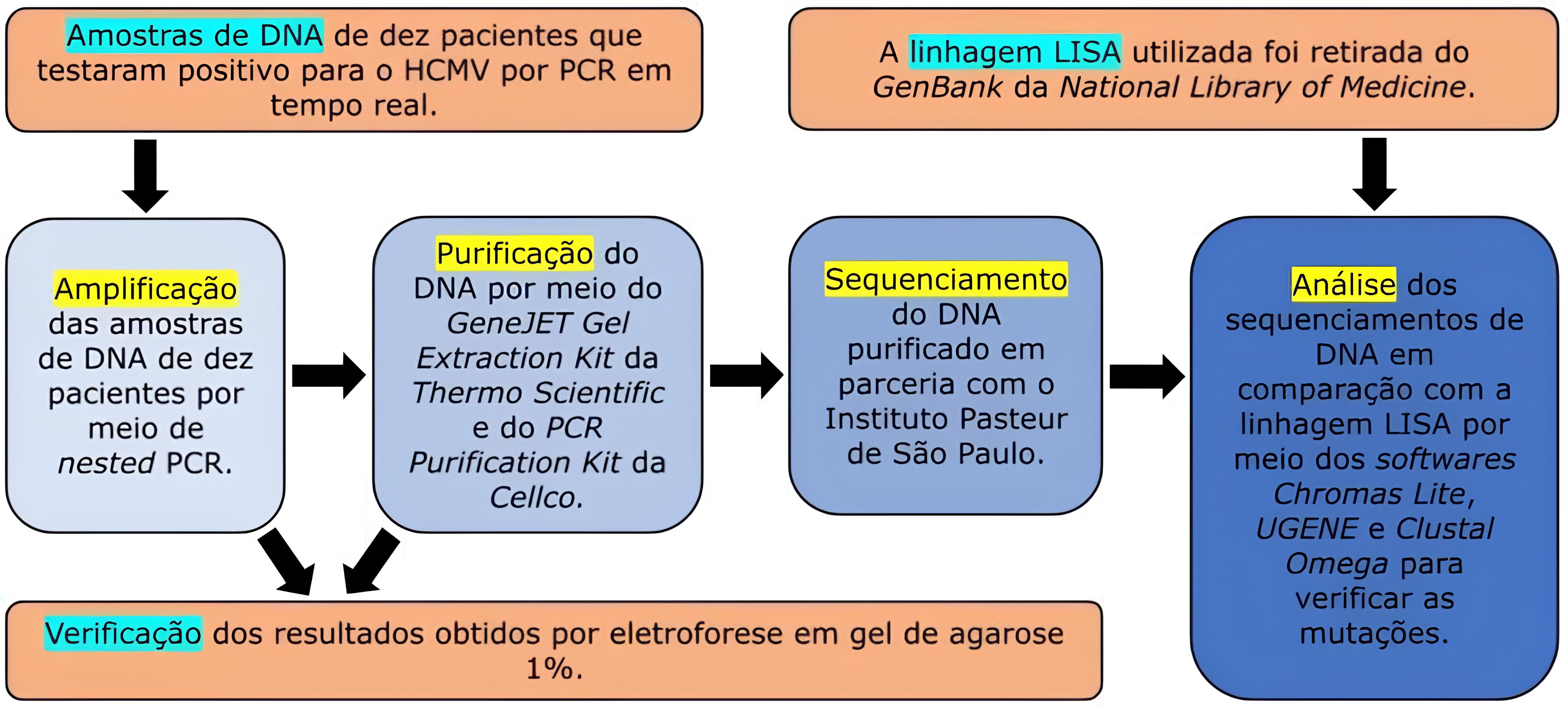
Figura 2. O caminho utilizado na busca dos resultados. Elaborado pelas autoras.
O que foi encontrado? O que pode significar?
Ao analisar o DNA do vírus na região UL111A presente nos dez pacientes e comparar com a região UL111A da linhagem LISA, foi possível visualizar diversas mutações. Entretanto, comparando os dez pacientes, foi identificada a presença de mutações similares. Essas semelhanças são apresentadas na Tabela 1.
Tabela 1. Mutações nos nucleotídeos semelhantes nos dez pacientes. Elaborado pelas autoras.
|
Pacientes |
Nucleotídeo e mutação na base nitrogenada |
|
03 |
79/80 - Adição de trinca (CGA) |
|
04 |
81/82 - Adição de trinca (ACG) |
|
01, 02, 05, 06, 07, 08, 09 e 10 |
299 – A para G |
|
03 e 04 |
302 – A para G |
|
01, 02, 05, 09 e 10 |
328 – T para C |
|
03 e 04 |
331 – T para C |
|
06, 07, 08 e 09 |
397 – T para C |
|
05 e 10 |
379 – C para T |
Analisando a Tabela 1, foi possível notar que os pacientes 01, 02, 05, 06, 07, 08, 09 e 10 apresentaram uma mutação no nucleotídeo 299. Essa variação também foi verificada nos pacientes 03 e 04, porém no nucleotídeo 302, pois houve a adição de uma trinca em uma variação anterior. Dessa forma, foi possível notar que os dez pacientes apresentaram a mesma mutação. Também foi possível notar que os pacientes 01, 02, 05, 09 e 10 apresentaram uma mutação no nucleotídeo 328. Essa variação também foi percebida nos pacientes 03 e 04, porém no nucleotídeo 331, novamente por adição da trinca. Assim, sete dos dez pacientes apresentaram essa mesma mutação. Além disso, foi possível notar que os pacientes 06, 07, 08 e 09 mostraram possuir a mesma mutação no nucleotídeo 397. Sendo assim, quatro de dez pacientes apresentaram essa mutação. Os pacientes 05 e 10 apresentaram a mesma mutação no nucleotídeo 379. Assim, dois de dez pacientes apresentaram essa mutação. Os pacientes 03 e 04 apresentaram a adição de uma trinca em seu DNA, ou seja, houve a adição de três nucleotídeos. O paciente 03 apresentou a adição da trinca CGA entre os nucleotídeos 79 e 80. O paciente 04 apresentou a adição da trinca ACG entre os nucleotídeos 81 e 82.
Por meio das tecnologias utilizadas, foi possível utilizar o DNA de cada um dos pacientes com suas respectivas mutações e simular, a partir deles, os aminoácidos que seriam produzidos. Ao realizar esse processo, foi possível notar que houve mutações também nos aminoácidos, em comparação com os aminoácidos formados a partir da UL111A LISA. As mutações semelhantes são apresentadas na Tabela 2.
Tabela 2. Mutações nos aminoácidos semelhantes nos dez pacientes. Elaborado pelas autoras.
|
Pacientes |
Nucleotídeo e mutação na base nitrogenada |
Aminoácido e mutação |
|
03 |
79/80 - Adição de trinca (CGA) |
28/29 - Adição de uma Treonina |
|
04 |
81/82 - Adição de trinca (ACG) |
28/29 - Adição de uma Treonina |
|
01, 02, 05, 06, 07, 08, 09 e 10 |
299 – A para G |
98 – Aspartato para Glicina |
|
03 e 04 |
302 – A para G |
98 – Aspartato para Glicina |
|
01, 02, 05, 09 e 10 |
328 – T para C |
108 – Tirosina para Histidina |
|
03 e 04 |
331 – T para C |
108 – Tirosina para Histidina |
|
06, 07, 08 e 09 |
397 – T para C |
131 – Stop códon para Glutamina |
|
05 e 10 |
379 – C para T |
125 – Prolina para Serina |
Analisando a Tabela 2, foi possível notar que todos os dez pacientes apresentaram uma mutação no aminoácido 98 em comparação com a UL111A LISA. Também foi possível notar que os pacientes 01, 02, 03, 04, 05, 09 e 10 apresentaram uma mutação no aminoácido 108 em comparação com a UL111A LISA. Assim, sete dos dez pacientes apresentaram essa mesma variação. Além disso, foi possível notar que os pacientes 06, 07, 08 e 09 mostraram possuir uma mutação no aminoácido 131 em comparação com a UL111A LISA. Sendo assim, quatro de dez pacientes apresentaram essa variação. Os pacientes 05 e 10 apresentaram a mesma mutação no aminoácido 125 em comparação com a UL111A LISA. Verificou-se que dois de dez pacientes apresentaram essa variação. Por fim, os pacientes 03 e 04 apresentaram a adição de uma treonina entre os aminoácidos 28 e 29 em comparação com a UL111A LISA.
Até aqui, foram abordados os nucleotídeos e os aminoácidos, mas e as proteínas? Lembrando que a parte UL111A do Citomegalovírus pode produzir oito proteínas diferentes4 5 6, sendo que duas delas possuem as mesmas propriedades da IL-10, que é a proteína que o corpo humano produz e o vírus copia. Colocando essas duas proteínas em foco (cmvIL-104 e LAcmvIL-105), foi possível notar que os dez pacientes apresentaram uma mesma mutação no aminoácido 75 nessas duas proteínas. Essa variação foi fruto da mutação no nucleotídeo 299 e resultou na modificação de uma Metionina para uma Valina.
Tá, mas qual foi a mutação mais importante e o que ela pode afetar?
Apesar de ocorrerem diversas mutações na região UL111A do DNA do Citomegalovírus nas amostras dos dez pacientes, foi possível concluir que oito dos dez pacientes estudados apresentaram a mesma mutação no nucleotídeo 299, uma modificação de A para G. Essa mutação também foi percebida nos outros dois pacientes, porém no nucleotídeo 302, pois houve a adição de uma trinca em uma variação anterior. Assim, concluiu-se que os dez pacientes apresentaram a mesma mutação. Ela foi responsável pela mutação no aminoácido 98 em comparação com a região UL111A do HCMV da linhagem LISA. Essa mutação deu origem a uma variação no aminoácido 75 nas duas proteínas produzidas pelo vírus que possuem as mesmas propriedades da IL-10, produzida pelo ser humano. O resultado foi a modificação de uma Metionina para uma Valina nos dez pacientes estudados.
Essas mutações encontradas podem possibilitar que o vírus adquira outras capacidades de se proliferar, de infectar o hospedeiro e de permanecer nele. Diante desse resultado, mais estudos são necessários com o intuito de verificar se essas duas proteínas produzidas pelo vírus possuem modificações em sua função quando apresentam essa mutação. Além disso, mais estudos podem ser realizados com o objetivo de investigar se as outras mutações presentes nos pacientes apresentam relação com a doença identificada nesses indivíduos, ou seja, com a perda renal. A expectativa é de que esses estudos possam levar ao desenvolvimento de novos tratamentos para pessoas acometidas com deficiência nos rins, o que pode chegar a ser uma inovação nessa área, com a possibilidade de aumento do tempo de vida dos pacientes.
Vale lembrar que esse estudo é apenas um ponto de partida. Aqui, foram analisadas apenas 10 amostras de pacientes do Hospital do Rim de São Paulo. É necessário que o mesmo estudo seja replicado com um número maior de pacientes e localizados em diversas regiões, com a finalidade de aumentar o número de amostras e permitir que padrões sejam visualizados e estudados. Além disso, outras pesquisas semelhantes podem ser realizadas com pessoas que apresentam outras enfermidades, com o objetivo de possibilitar o desenvolvimento de novas formas de tratamento dessas doenças.
O nosso “Muito Obrigada!”
Grande agradecimento ao Instituto Pasteur de São Paulo, à Giovana Carneiro, à Isabella Martines, ao Wemenes Silva e a todos os pesquisadores do Laboratório de Bioquímica Molecular da UFABC. Acima de tudo, à Universidade Federal do ABC por todos os equipamentos, materiais, espaço e conhecimentos concedidos.
Referências
1. Collins-McMillen, D., Buehler, J., Peppenelli, M., & Goodrum, F. Molecular determinants and the regulation of human cytomegalovirus latency and reactivation. Viruses. 10(8), 444 (2018).
2. Redpath, S., Ghazal, P., & Gascoigne, N. R. Hijacking and exploitation of IL-10 by intracellular pathogens. Trends in microbiology. 9(2), 86-92, (2001).
3. Eskdale, J., Kube, D., Tesch, H., & Gallagher, G. Mapping of the human IL10 gene and further characterization of the 5’flanking sequence. Immunogenetics. 46, 120-128, (1997).
4. Waters, S., Lee, S., Ariyanto, I., Kresoje, N., Leary, S., Munyard, K., Gaudieri, S., Irish, A., Keil, A. D., Allcock, R. J. N. & Price, P. Sequencing of the viral UL111a gene directly from clinical specimens reveals variants of HCMV-encoded IL-10 that are associated with altered immune responses to HCMV. International Journal of Molecular Sciences. 23(9), 4644, (2022).
5. Lin, Y. L., Chang, P. C., Wang, Y., & Li, M. Identification of novel viral interleukin-10 isoforms of human cytomegalovirus AD169. Virus research. 131(2), 213-223, (2008).
6. Pantaleão, S. Q., Camillo, L. D. M. B., Neves, T. C., Menezes, I. D. G., Stangherlin, L. M., Batista, H. B. D. C. R., Poole, E., Nevels, M., Philot, E. A., Scott, A. L. & da Silva, M. C. C. Molecular modelling of the HCMV IL-10 protein isoforms and analysis of their interaction with the human IL-10 receptor. Plos one. 17(11), e0277953, (2022).
7. Cunningham, C., Gatherer, D., Hilfrich, B., Baluchova, K., Dargan, D. J., Thomson, M., Griffiths, P. D., Wilkinson, G. W. G., Schulz, T. F. & Davison, A. J. Sequences of complete human cytomegalovirus genomes from infected cell cultures and clinical specimens. The Journal of general virology. 91(Pt 3), 605, (2010).
8. Murphy, E., Yu, D., Grimwood, J., Schmutz, J., Dickson, M., Jarvis, M. A., Hahn, G., Nelson, J. A., Myers, R. M. & Shenk, T. E. Coding potential of laboratory and clinical strains of human cytomegalovirus. Proceedings of the National Academy of Sciences. 100(25), 14976-14981, (2003).
9. Garrigue, I., Faure-Della Corte, M., Magnin, N., Couzi, L., Capdepont, S., Rio, C., Merville, P., Dechanet-Merville, J., Fleury, H. & Lafon, M. E. Variability of UL18, UL40, UL111a and US3 immunomodulatory genes among human cytomegalovirus clinical isolates from renal transplant recipients. Journal of clinical virology. 40(2), 120-128, (2007).
10. Sijmons, S., Thys, K., Mbong Ngwese, M., Van Damme, E., Dvorak, J., Van Loock, M., Guangdi, L., Tachezy, R., Busson, L., Aerssens, J., Ranst, M. V. & Maes, P. High-throughput analysis of human cytomegalovirus genome diversity highlights the widespread occurrence of gene-disrupting mutations and pervasive recombination. Journal of virology. 89(15), 7673-7695, (2015).

Redes Sociais